

(Français)
The goal of PlotFTIR is to easily and quickly kick-start
the production of journal-quality Fourier Transform Infra-Red (FTIR)
spectral plots in R using ggplot2. The produced plots can be published
directly or further modified by ggplot2 functions.
You can install the development version of PlotFTIR from
GitHub with:
# install.packages("devtools")
devtools::install_github("NRCan/PlotFTIR")
library(PlotFTIR)This is a basic example which shows you how to plot a prepared set of FTIR spectra:
biodiesel_plot <- plot_ftir(biodiesel)
biodiesel_plot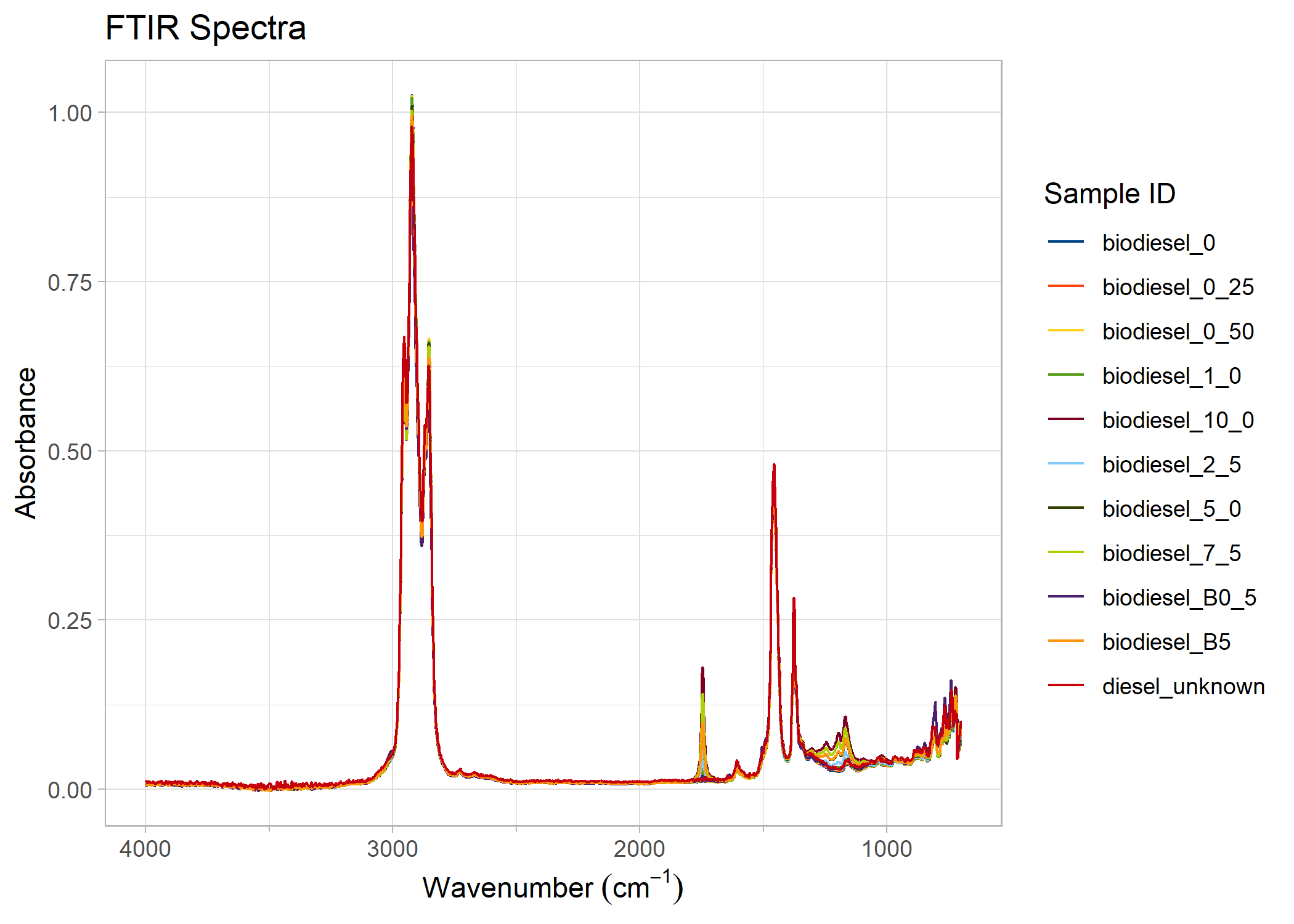
You can also plot spectra in a stacked/offset manner instead of overlaid:
# Generate a plot
plot_ftir_stacked(biodiesel)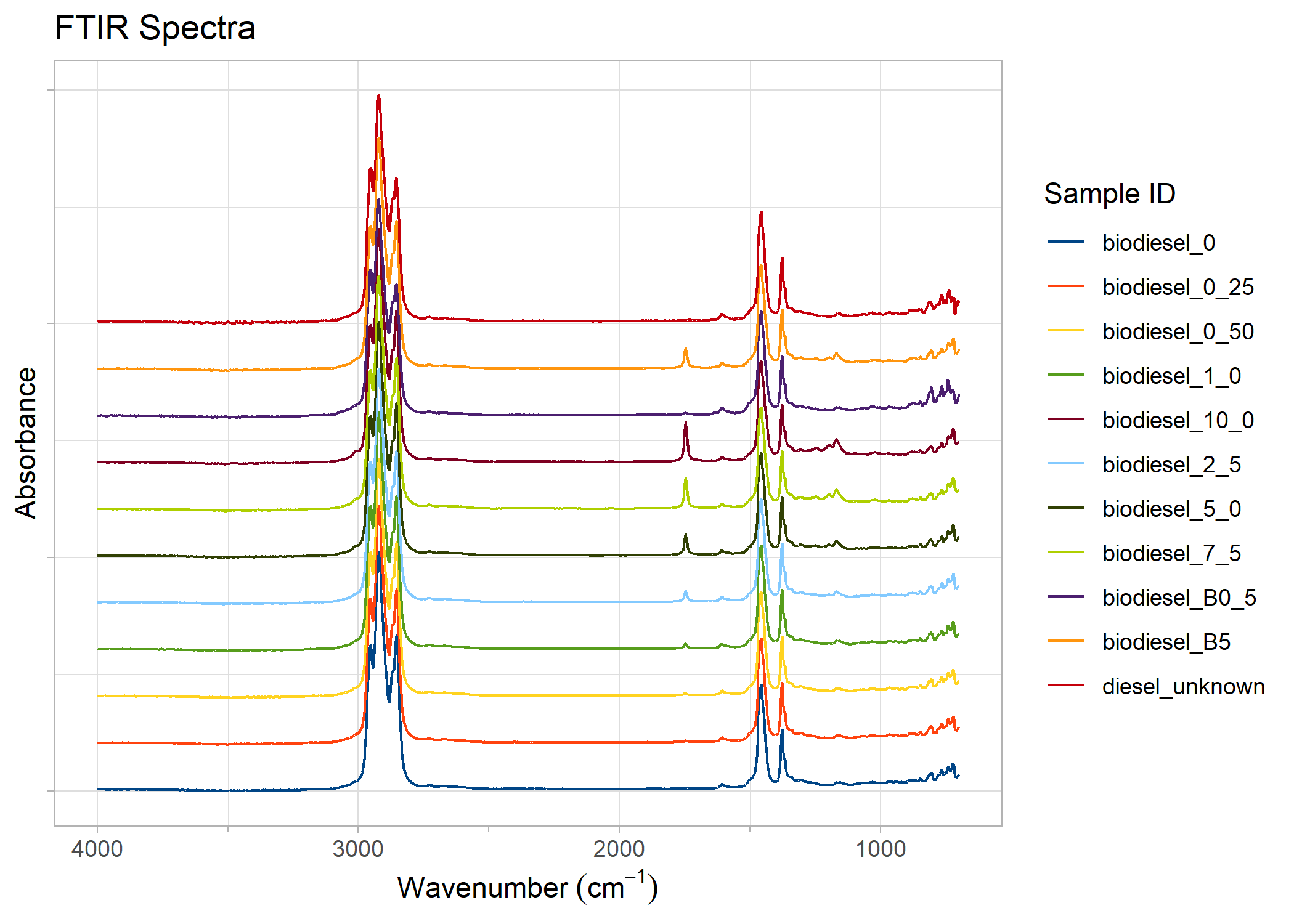
Note the default plot and legend titles are in english but can be
automatically changed to french defaults by supplying the
lang = 'fr' argument to plot creation functions.
Plots can be manipulated, for example, by zooming in on a range:
# Zoom to a specified range of 1850 to 1650 cm^-1
zoom_in_on_range(biodiesel_plot, c(1650, 1850))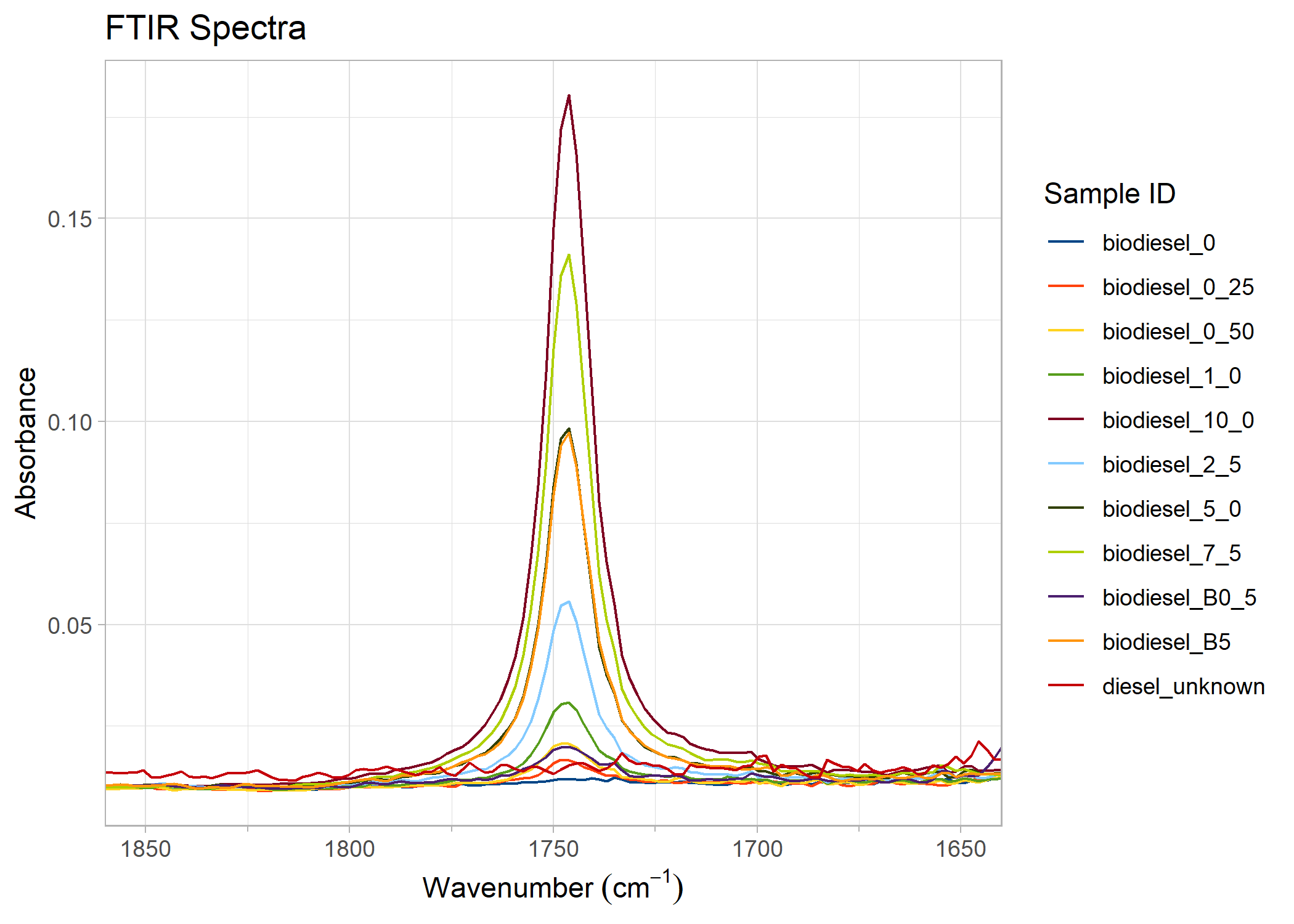
Some FTIR plots have a compressed low-energy portion of the graph which you might wish to zoom in on. You can achieve this by the following:
# compress the data with wavenumbers above 2000 (to the left of 2000 on the
# plot) by a factor of 5
compress_low_energy(biodiesel_plot, cutoff = 2000, compression_ratio = 5)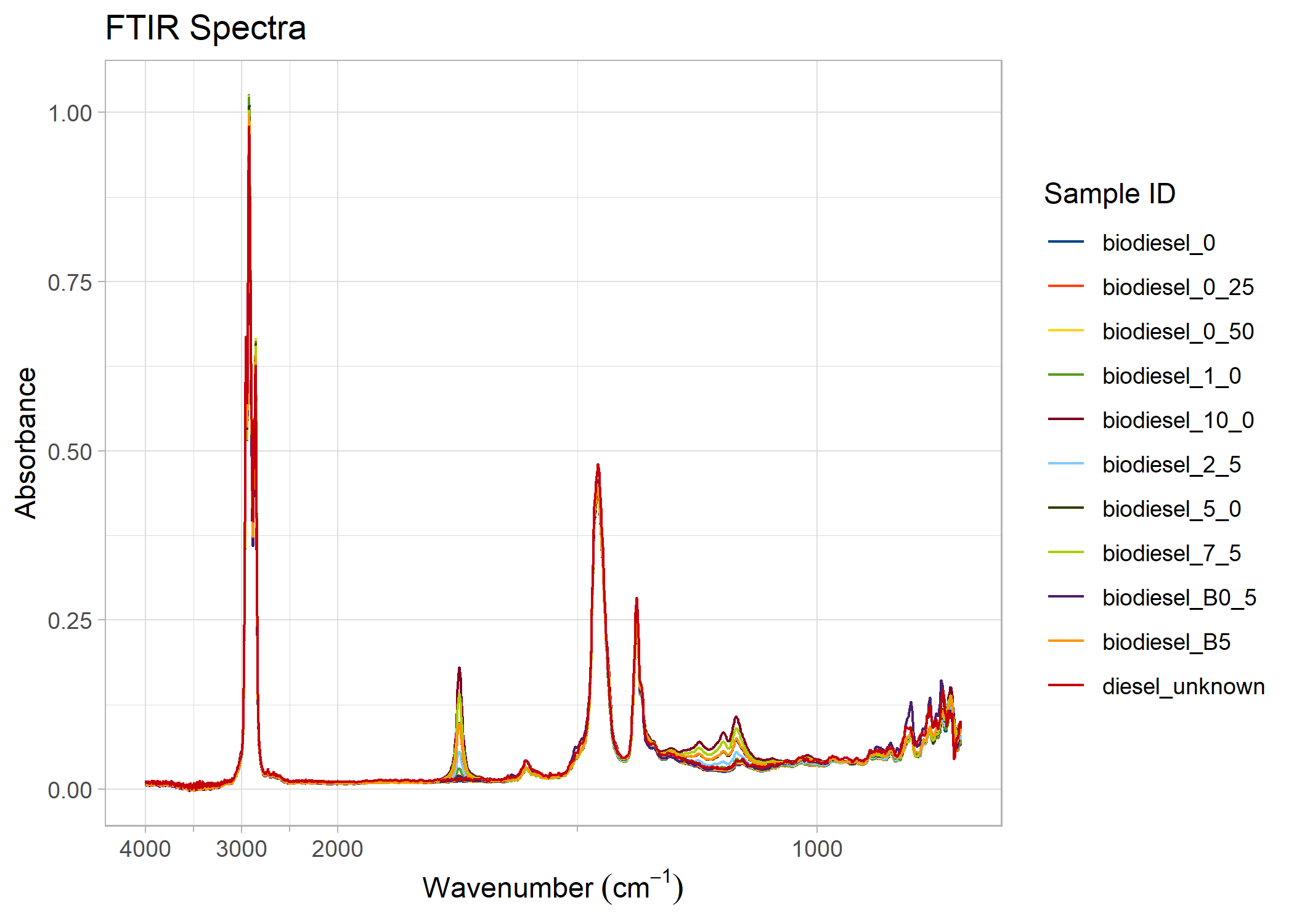
You can also add marker lines (with labels) at specific wavenumbers on the plots, controlling their line or text properties as needed. Similarly, a shaded band can be added to indicate a region.
biodiesel_marked <- add_wavenumber_marker(biodiesel_plot,
wavenumber = 1742,
text = "C=O Stretch",
label_aesthetics = list("color" = "red")
)
add_band(biodiesel_marked, c(2750,3050), "C-H Stretch")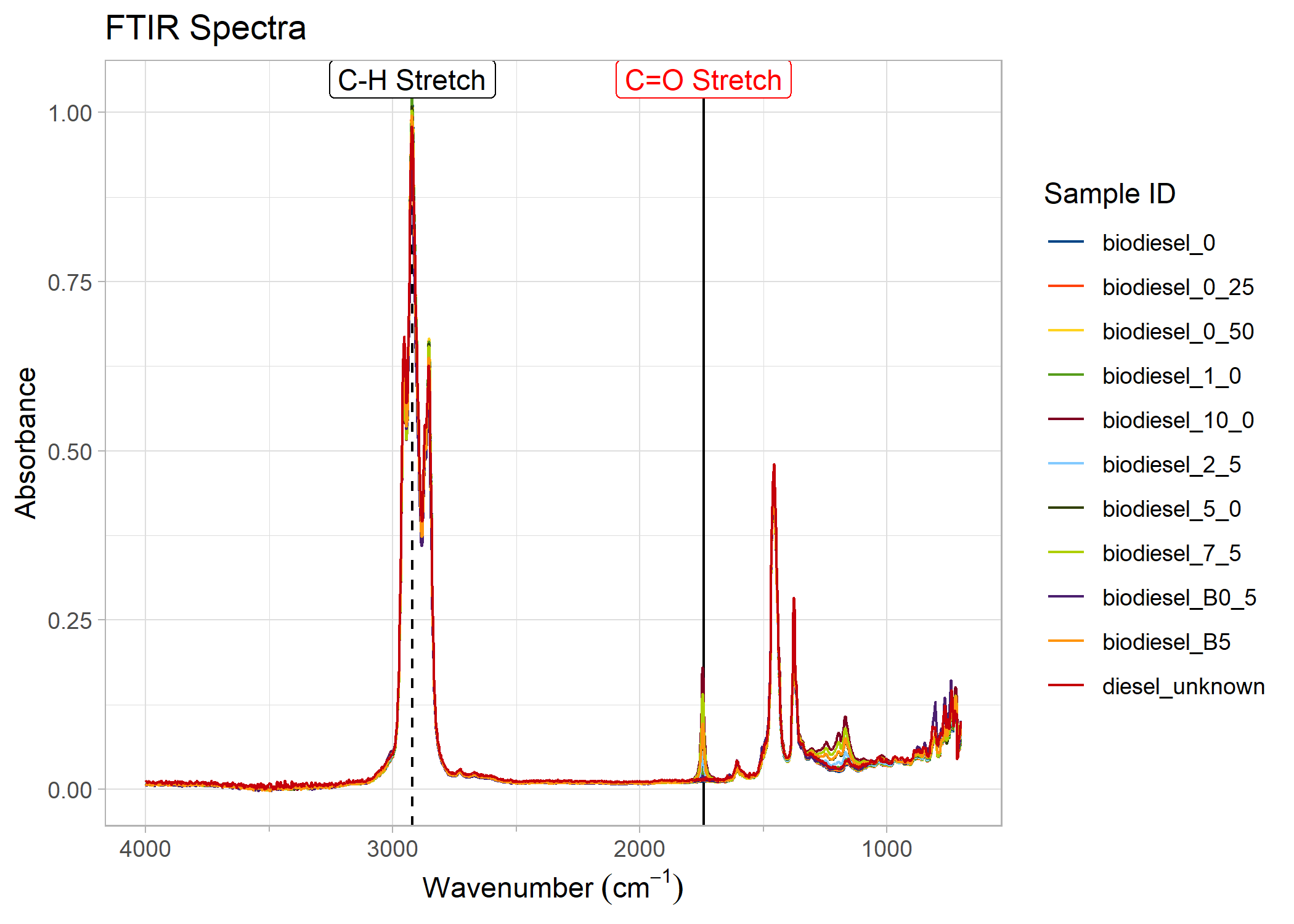
If the need arises to rename samples listed in the legend, this is
possible via rename_plot_sample_ids(). Samples must be
listed in the rename vector with the format
"new name" = "old name".
new_names <- c(
"0.0% Biodiesel" = "biodiesel_0",
"0.25% Biodiesel" = "biodiesel_0_25",
"0.50% Biodiesel" = "biodiesel_0_50",
"1.0% Biodiesel" = "biodiesel_1_0",
"2.5% Biodiesel" = "biodiesel_2_5",
"5.0% Biodiesel" = "biodiesel_5_0",
"7.5% Biodiesel" = "biodiesel_7_5",
"10.0% Biodiesel" = "biodiesel_10_0",
"Commercial B0.5" = "biodiesel_B0_5",
"Commercial B5" = "biodiesel_B5",
"Unknown Biodiesel" = "diesel_unknown"
)
rename_plot_sample_ids(biodiesel_plot, new_names)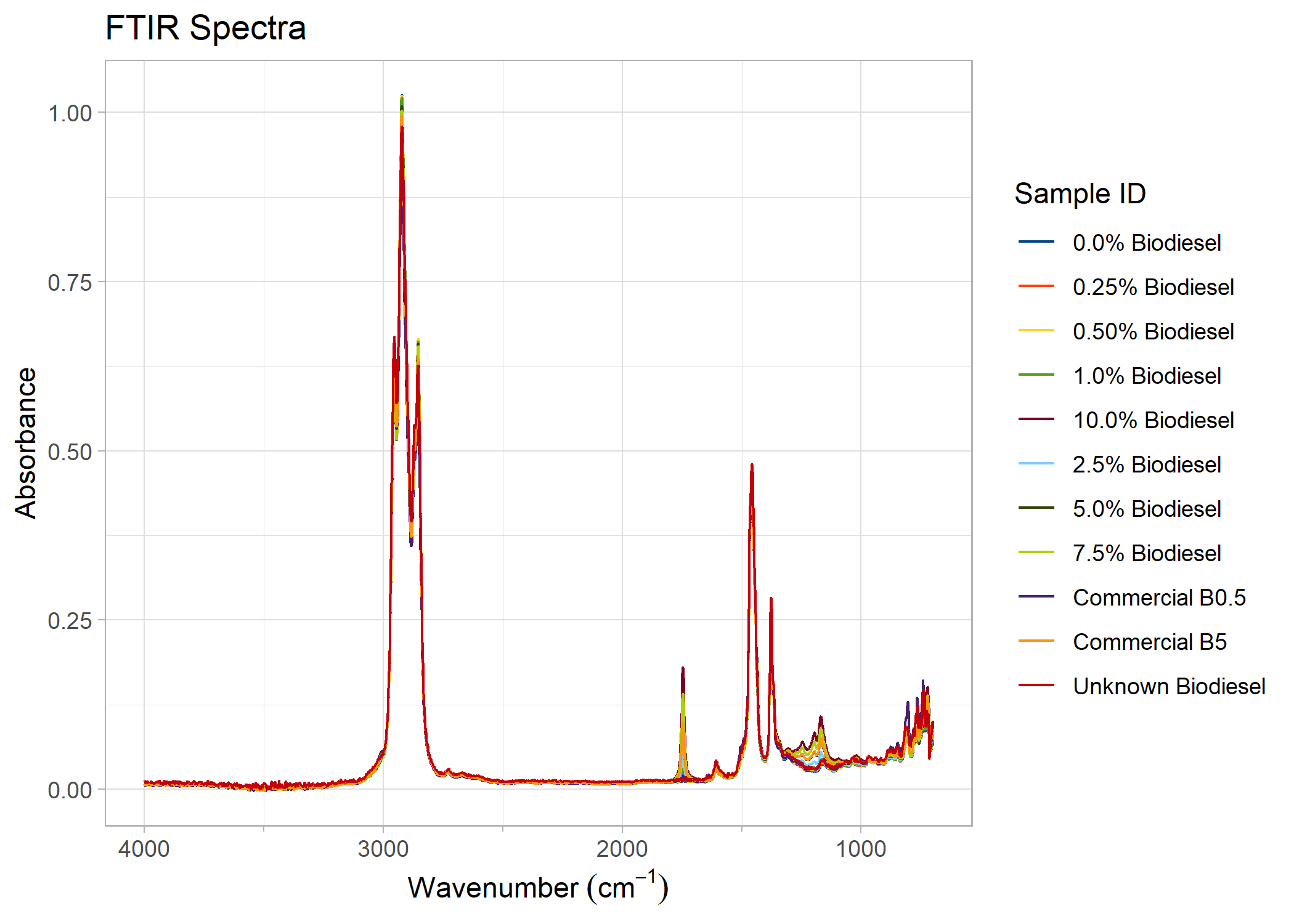
A helper function for the renaming is provided (see the documentation
for get_plot_sample_ids()).
Specific sample(s) can be highlighted (other samples greyed out) by
calling highlight_sample().
Finally, plot legends are customizable (for basic changes) through a
helper function move_plot_legend().
The package contains two datasets to provide example spectra for
plotting: * biodiesel is a set of diesels with 0 to 10 %
FAMEs (fatty acid methyl esters; biodiesel) content, plus two known and
one unknown diesel spectra. * sample_spectra is a set of
random FTIR spectra which includes spectra of pure toluene, isopropanol,
and heptanes, as well as white printer paper and a polystyrene film.
An example of the biodiesel data set is below:
head(biodiesel)
#> wavenumber absorbance sample_id
#> 1 700.7395 0.072530 biodiesel_0
#> 2 702.6032 0.065398 biodiesel_0
#> 3 704.4669 0.063371 biodiesel_0
#> 4 706.3305 0.059454 biodiesel_0
#> 5 708.1942 0.058133 biodiesel_0
#> 6 710.0579 0.056636 biodiesel_0Note that because most functions return a data type similar to what
is provided, tidy-eval is possible (using the base R pipe
|> or magrittr
pipe function %>%).
library(magrittr)
new_ids <- c(
"Toluene" = "toluene", "C7 Alkane" = "heptanes", "IPA" = "isopropanol",
"White Paper" = "paper", "PS Film" = "polystyrene"
)
sample_spectra |>
absorbance_to_transmittance() |>
plot_ftir(plot_title = "Example FTIR Spectra") |>
zoom_in_on_range(zoom_range = c(3800, 800)) |>
compress_low_energy(compression_ratio = 4) |>
add_wavenumber_marker(
wavenumber = 1495,
text = "C-C Aromatic",
line_aesthetics = list("linetype" = "dashed"),
label_aesthetics = list("color" = "#7e0021")
) |>
add_wavenumber_marker(
wavenumber = 3340,
text = "O-H Alcohol",
line_aesthetics = list("linetype" = "dotted"),
label_aesthetics = list("color" = "#ff420e")
) |>
rename_plot_sample_ids(sample_ids = new_ids) |>
move_plot_legend(position = "bottom", direction = "horizontal")
#> Coordinate system already present. Adding new coordinate system, which will
#> replace the existing one.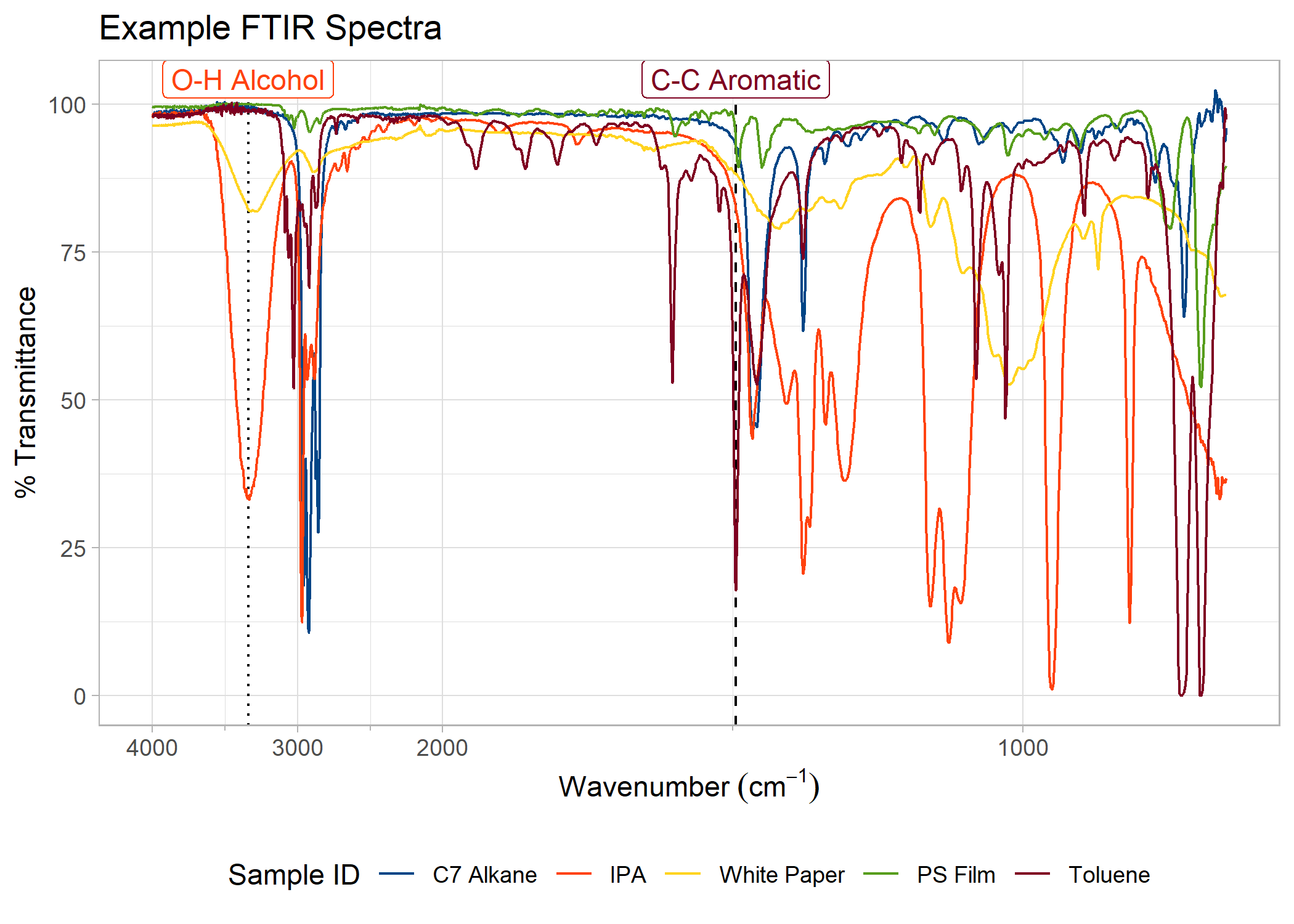
FTIR spectral data can be converted between absorbance and
transmittance. Only one type of data can exist in a data.frame and be
plotted. The functions absorbance_to_transmittance() and
transmittance_to_absorbance() perform these
conversions.
biodiesel_transm <- absorbance_to_transmittance(biodiesel)
head(biodiesel_transm)
#> wavenumber transmittance sample_id
#> 1 700.7395 84.61941 biodiesel_0
#> 2 702.6032 86.02051 biodiesel_0
#> 3 704.4669 86.42293 biodiesel_0
#> 4 706.3305 87.20593 biodiesel_0
#> 5 708.1942 87.47159 biodiesel_0
#> 6 710.0579 87.77362 biodiesel_0Functions are provided for adjusting the baseline of spectra, adding
or subtracting scalar values from entire spectra, normalizing spectra,
and averaging spectra, see: * recalculate_baseline() *
add_scalar_value() and subtract_scalar_value()
* normalize_spectra() * average_spectra()
PlotFTIR can read .csv, .asp, and .jdx file types. The
.csv or .jdx files should contain only one spectra, with columns for
wavenumber and absorbance or
transmittance. The .asp files should be according to the
file specifications (not modified by the user).
ir and ChemoSpec PackagesPlotFTIR has functions to interface with the
ir package by Henning Teickner. This package offers complex
baseline capabilities, smoothing, and more data analysis tools. More
information on the ir package is available in their documetation (via
CRAN). There is also capabilities to interface with
ChemoSpec package by Bryan Hanson, which supports advanced
statistics and chemometrics of spectral data. More information at the
ChemoSpec documentation.
Please cite this package in any journal articles containing images produced by way of the package. If installed from GitHub or CRAN the date field will be properly filled with the publishing year.
citation("PlotFTIR")
#> To cite package 'PlotFTIR' in publications use:
#>
#> Bulsink P (????). _PlotFTIR: Plot FTIR Spectra_. R package version
#> 1.1.0.9000, <https://github.com/NRCan/PlotFTIR>.
#>
#> A BibTeX entry for LaTeX users is
#>
#> @Manual{,
#> title = {PlotFTIR: Plot FTIR Spectra},
#> author = {Philip Bulsink},
#> note = {R package version 1.1.0.9000},
#> url = {https://github.com/NRCan/PlotFTIR},
#> }The package has the ability to change language from English to French
for plots on a per-plot basis (call plot_ftir() functions
with lang = 'en' or lang = 'fr arguments). In
addition, the default language can be set to English or French by
setting options('PlotFTIR.lang' = 'en') or
options('PlotFTIR.lang' = 'fr') respectively. This can be
added to your .RProfile to persist between R sessions.
(English)
Le but de PlotFTIR est de lancer facilement et
rapidement la production des tracés de spectres de spectroscopie
infrarouge à transformée de Fourier (IRTF) de qualité de revues
scientifiques dans le system R en utilisant ggplot2. Les tracés produits
peuvent être publiés directement ou modifiés par les fonctions
ggplot2.
Vous pouvez installer la version de développement de
PlotFTIR depuis GitHub
avec:
# install.packages("devtools")
devtools::install_github("NRCan/PlotFTIR")
library(PlotFTIR)Ceci est un example de base qui vous montre comment tracer un ensemble de spectres IRTF dejà preparé:
library(PlotFTIR)
plot_ftir(sample_spectra, lang = "fr")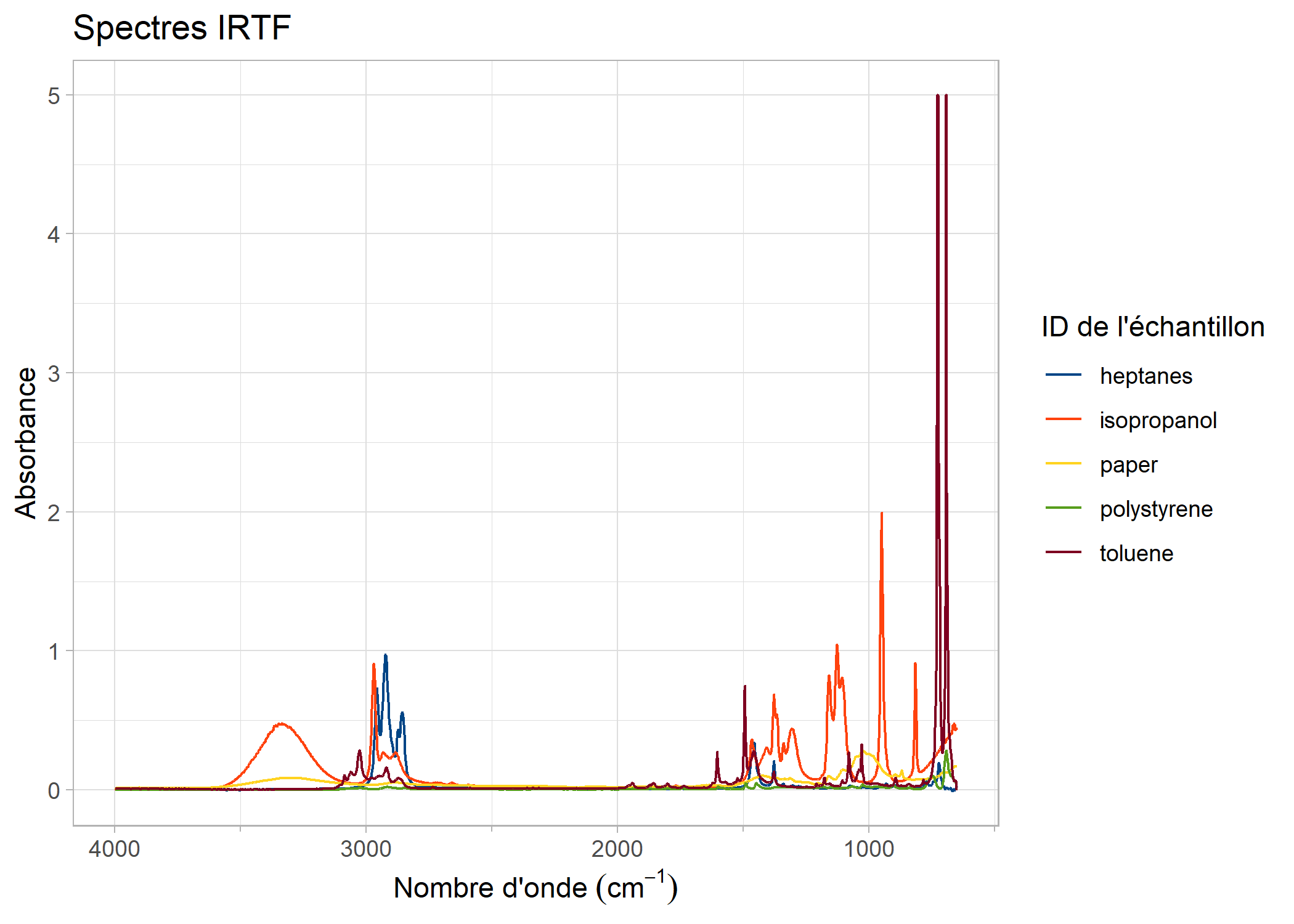
Vous pouvez également tracer les spectres de manière empilée/décalée au lieu de les superposer :
plot_ftir_stacked(biodiesel, plot_title = "Spectre IRTF empilée", lang = "fr")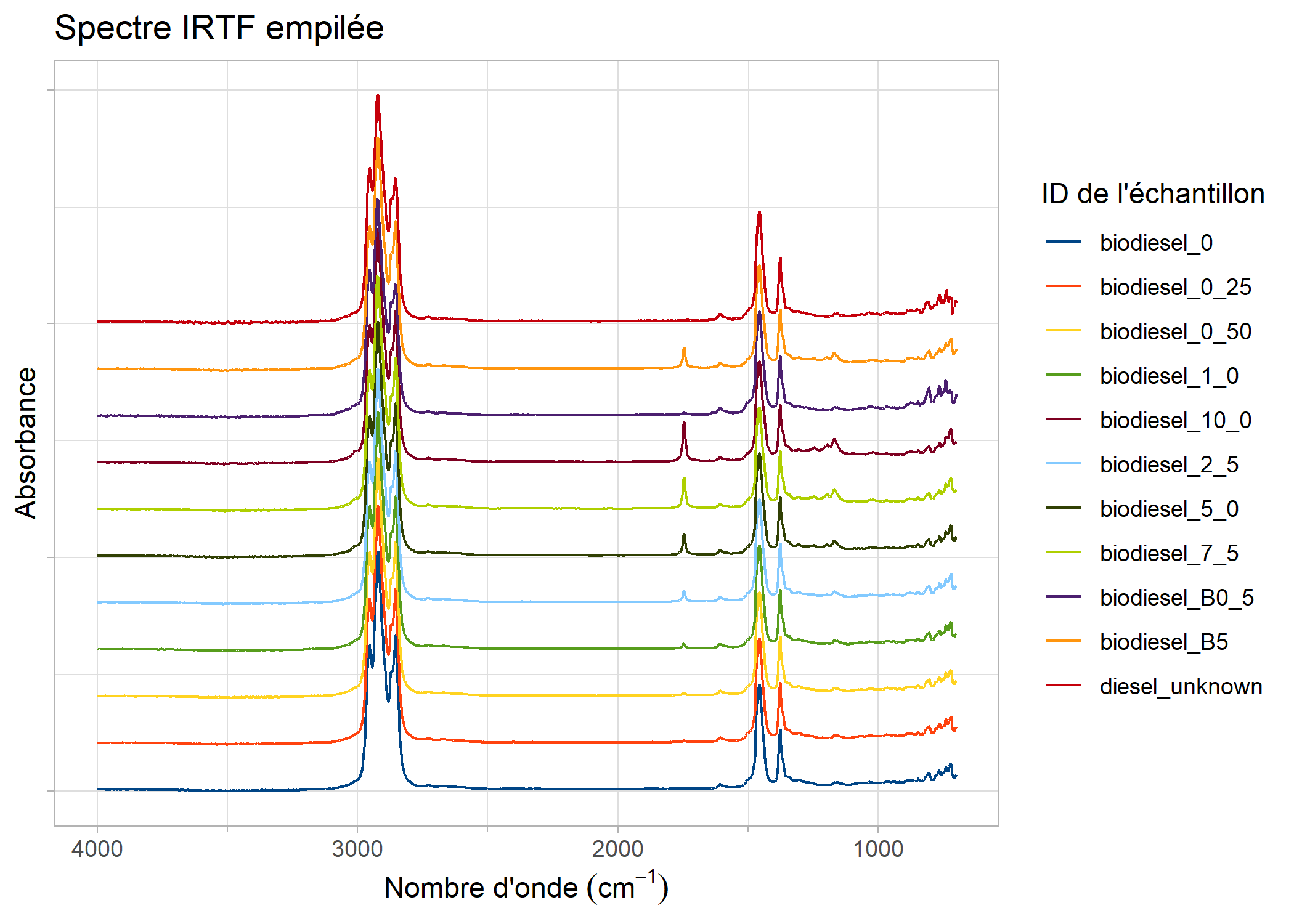
Notez que les titres par défaut de tracé et du légende sont en
anglais, mais qu’ils peuvent être automatiquement modifiés en français
en fournissant l’argument lang = 'fr' aux fonctions de
création de tracés.
Les tracés peuvent être manipulés, par exemple, en zoomant sur une plage :
# Générer un tracé
biodiesel_trace <- plot_ftir(biodiesel, lang = "fr")
# Zoom sur une plage spécifiée de 1850 à 1650 cm^-1
zoom_in_on_range(biodiesel_trace, c(1650, 1850))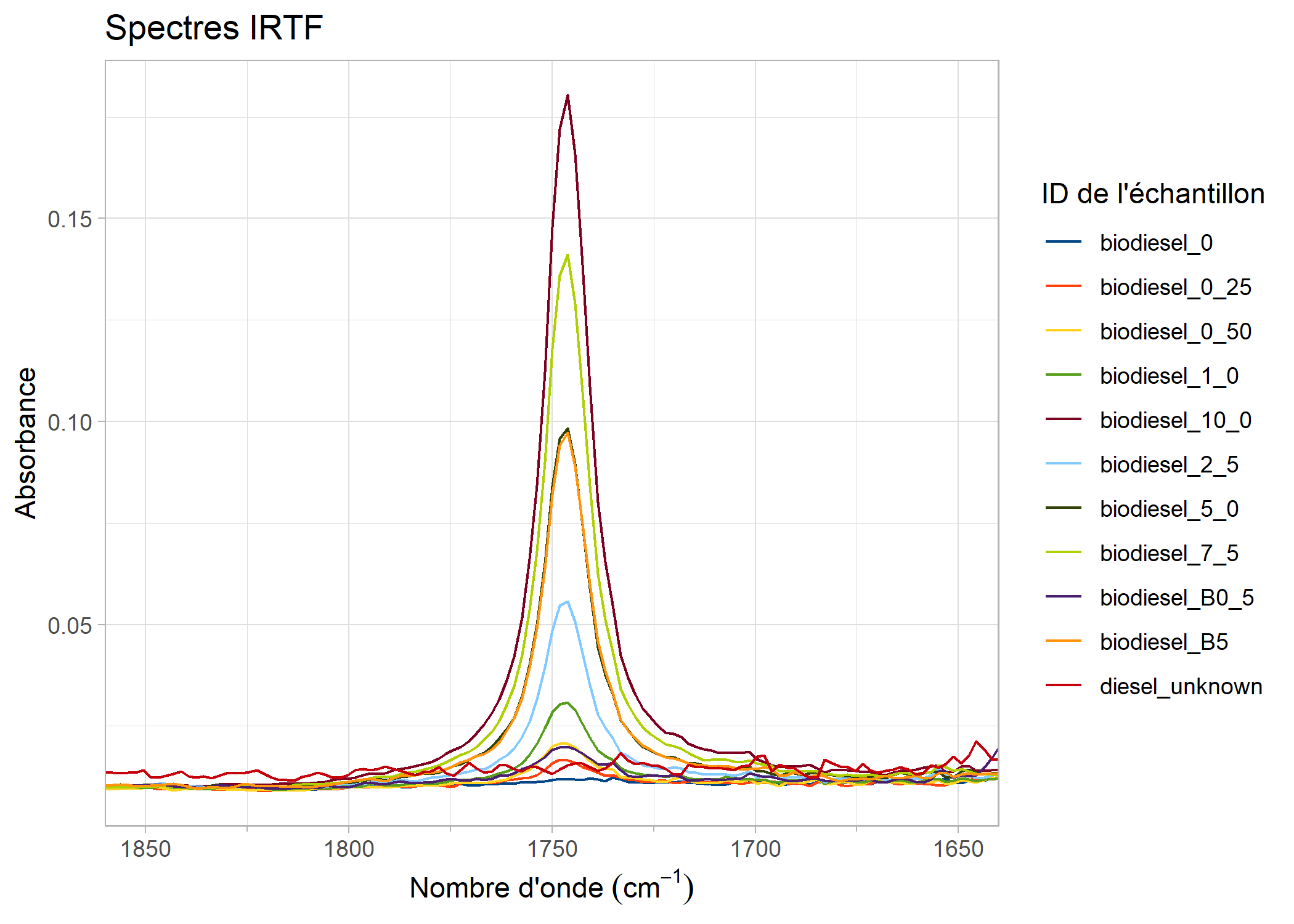
Certains tracés IRTF ont une partie compressée du graphique à faible énergie qui peuvent etre agrandie de la manière suivante :
# compresser les données avec des nombres d'onde supérieurs à 2000 (à gauche de
# 2000 sur le tracé) d'un facteur 5
compress_low_energy(biodiesel_trace, cutoff = 2000, compression_ratio = 5)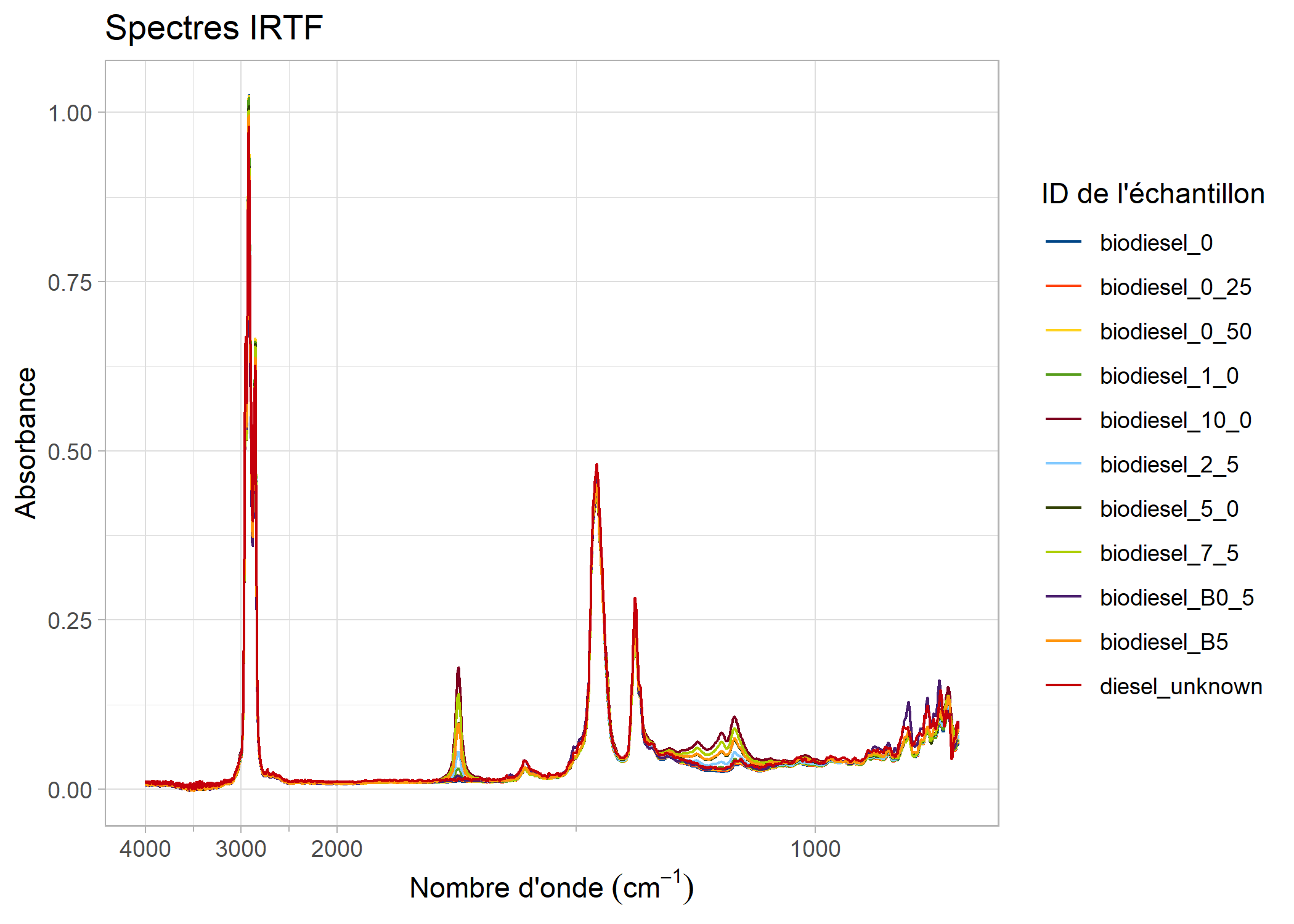
Vous pouvez également ajouter des lignes de marqueur (avec des étiquettes) à des numéros d’onde spécifiques sur les tracés, en contrôlant leurs propriétés de ligne ou de texte selon vos besoins. De même, une bande ombrée peut être ajoutée pour indiquer une région.
biodiesel_marked <- add_wavenumber_marker(biodiesel_trace,
wavenumber = 1742,
text = "C=O étirement",
label_aesthetics = list("color" = "red")
)
add_band(biodiesel_marked, c(2750,3050), "C-H étirement")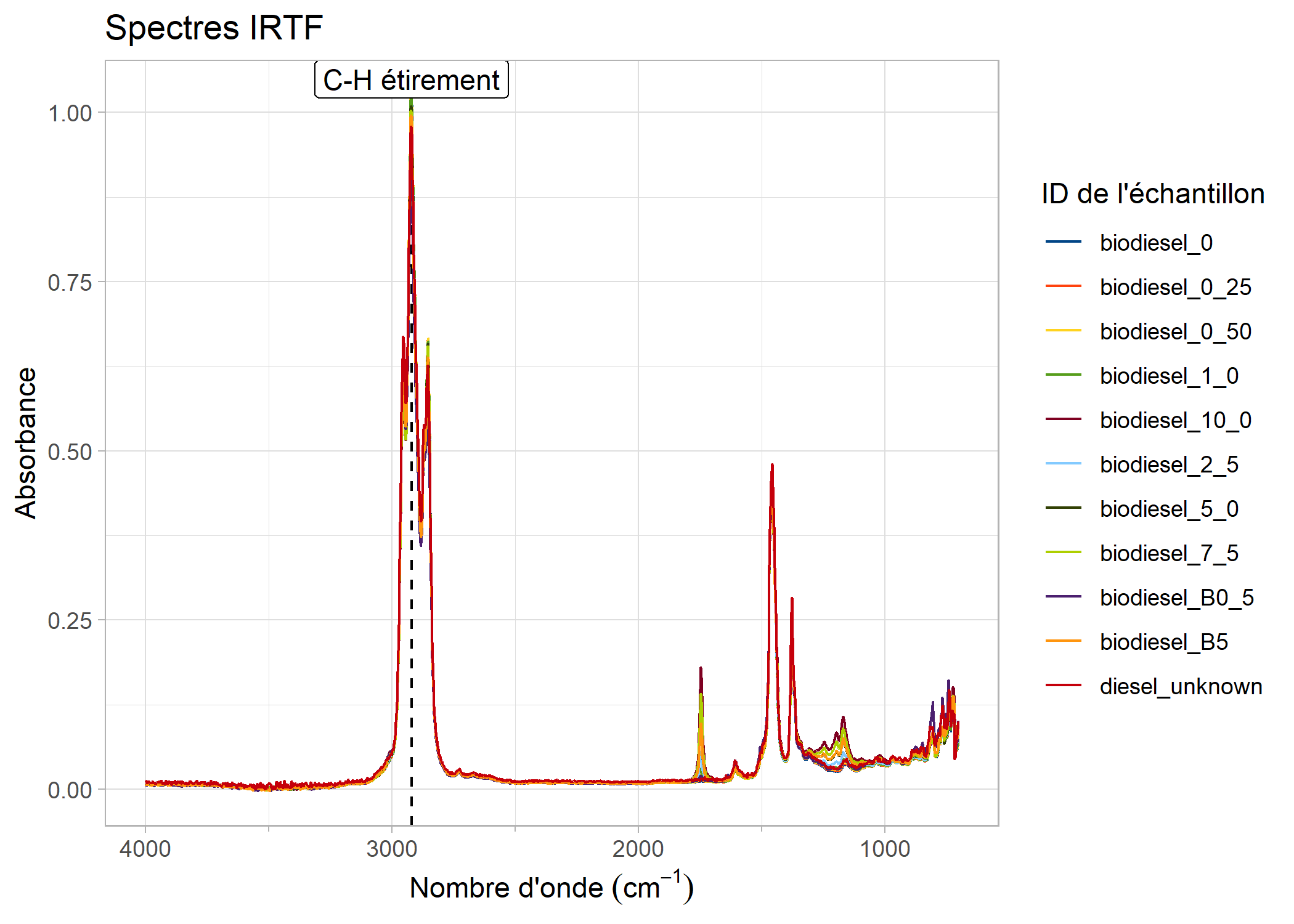
S’il est nécessaire de renommer les échantillons répertoriés dans la
légende, cela est possible via rename_plot_sample_ids(). Le
vecteur de renommage doit avoir le format
"nouveau nom" = "ancien nom".
nouveau_noms <- c(
"0,0% Biodiesel" = "biodiesel_0",
"0,25% Biodiesel" = "biodiesel_0_25",
"0,50% Biodiesel" = "biodiesel_0_50",
"1,0% Biodiesel" = "biodiesel_1_0",
"2,5% Biodiesel" = "biodiesel_2_5",
"5,0% Biodiesel" = "biodiesel_5_0",
"7,5% Biodiesel" = "biodiesel_7_5",
"10,0% Biodiesel" = "biodiesel_10_0",
"B0,5 Commercial" = "biodiesel_B0_5",
"B5 Commercial" = "biodiesel_B5",
"Biodiesel Inconnu" = "diesel_unknown"
)
rename_plot_sample_ids(biodiesel_trace, nouveau_noms)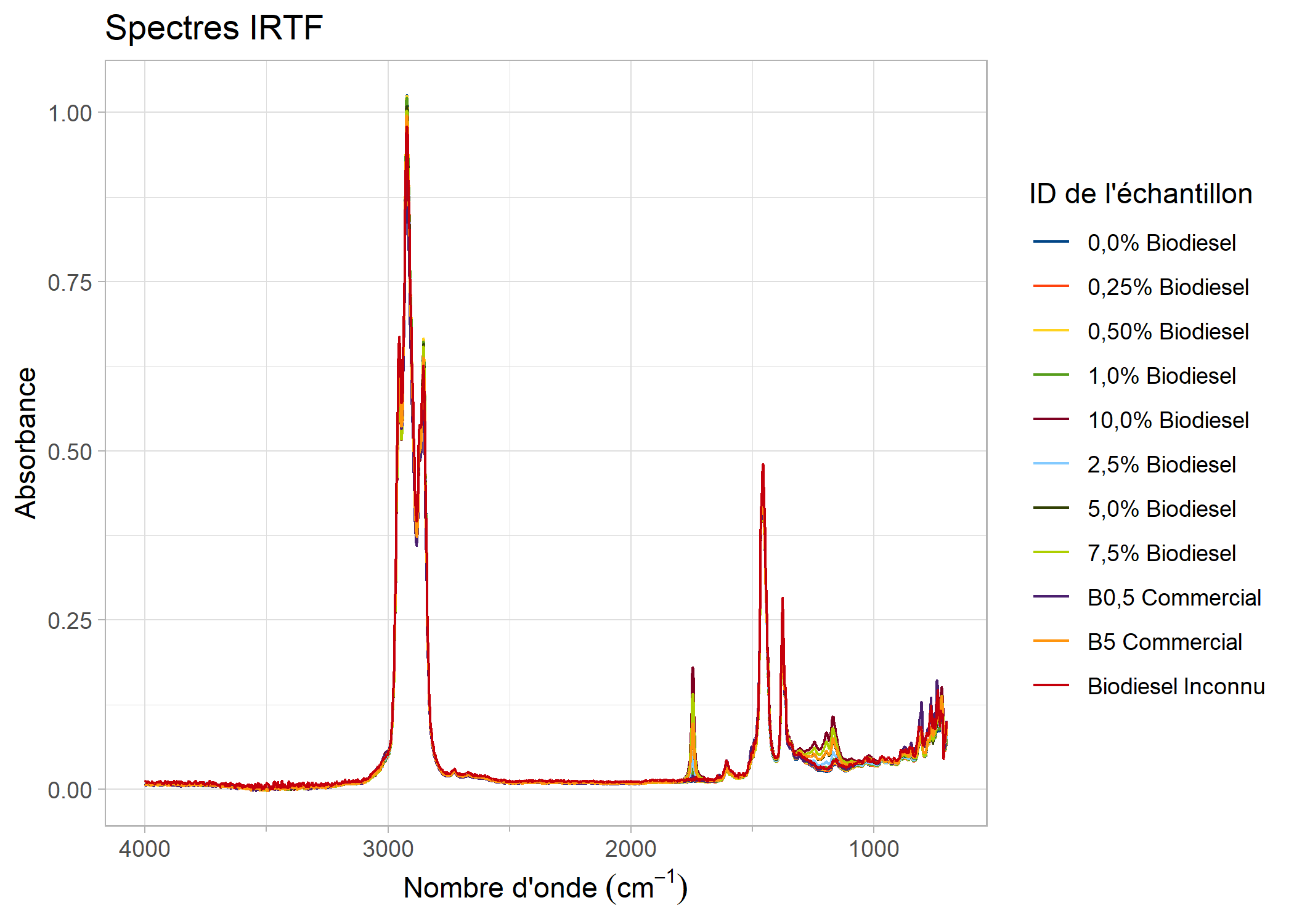
Une fonction d’assistance pour le changement de nom est fournie (voir
la documentation pour get_plot_sample_ids()).
Un ou plusieurs échantillons spécifiques peuvent être mis en évidence
(les autres échantillons étant grisés) en appelant
highlight_sample().
Enfin, les légendes des tracés sont personnalisables (pour les
modifications de base) via une fonction d’assistance
move_plot_legend().
Notez que comme la plupart des fonctions renvoient un type de données
similaire à celui qui est fourni, l’évaluation des données est possible
(en utilisant la
fonction magrittr tuyau %>%).
library(magrittr)
nouveaux_ids <- c(
"Toluène" = "toluene", "C7 alcane" = "heptanes", "alcool isopropylique" = "isopropanol",
"papier blanc" = "paper", "film de polystyrène" = "polystyrene"
)
sample_spectra |>
absorbance_to_transmittance() |>
plot_ftir(plot_title = "Exemple de spectres IRTF", lang = "fr") |>
zoom_in_on_range(zoom_range = c(3800, 800)) |>
compress_low_energy(compression_ratio = 4) |>
add_wavenumber_marker(
wavenumber = 1495,
text = "C-C aromatique",
line_aesthetics = list("linetype" = "dashed"),
label_aesthetics = list("color" = "#7e0021")
) |>
add_wavenumber_marker(
wavenumber = 3340,
text = "O-H alcool",
line_aesthetics = list("linetype" = "dotted"),
label_aesthetics = list("color" = "#ff420e")
) |>
rename_plot_sample_ids(sample_ids = nouveaux_ids) |>
move_plot_legend(position = "bottom", direction = "horizontal")
#> Coordinate system already present. Adding new coordinate system, which will
#> replace the existing one.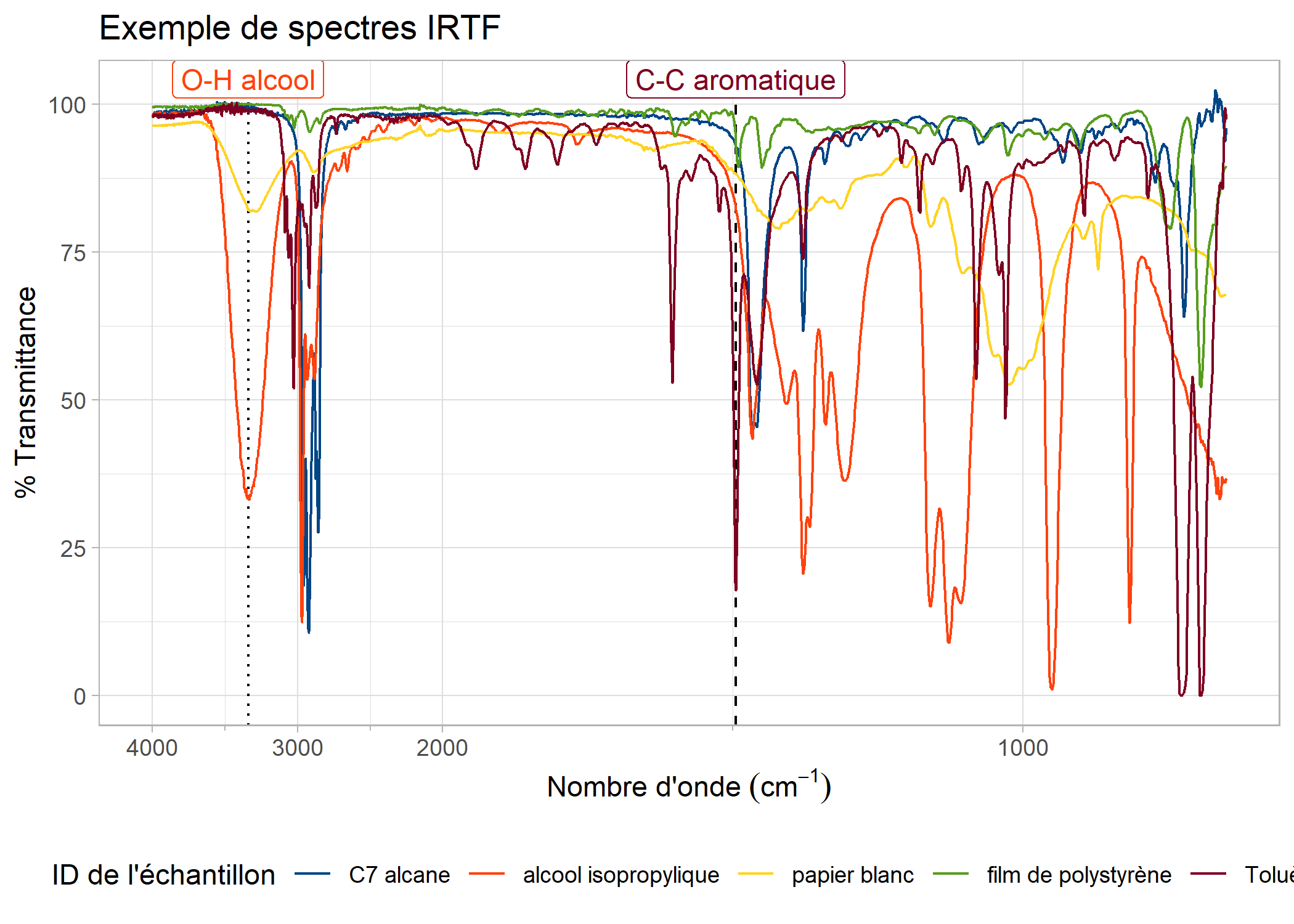
Notez que, comme la plupart des fonctions renvoient un type de données similaire à celui fourni, une “tidy-eval” est possible.
Le package contient deux ensembles de données pour fournir des
exemples de spectres à tracer: * biodiesel est un ensemble
de diesels avec une teneur en esters méthyliques d’acides gras (EMAGs)
(ou biodiesel) de 0 à 10 %, plus deux spectres de diesel connus et un
inconnu. * sample_spectra est un ensemble de spectres IRTF
aléatoires qui comprennent des spectres de toluène pur, d’isopropanol et
d’heptanes, ainsi que du papier d’imprimante blanc et un film de
polystyrène.
Un exemple de l’ensemble de données biodiesel est
ci-dessous:
head(biodiesel)
#> wavenumber absorbance sample_id
#> 1 700.7395 0.072530 biodiesel_0
#> 2 702.6032 0.065398 biodiesel_0
#> 3 704.4669 0.063371 biodiesel_0
#> 4 706.3305 0.059454 biodiesel_0
#> 5 708.1942 0.058133 biodiesel_0
#> 6 710.0579 0.056636 biodiesel_0Les données spectrales IRTF peuvent être converties entre
l’absorbance et la transmission. Un seul type de données peut exister
dans un data.frame et être tracé. Les fonctions
absorbance_to_transmittance() et
transmittance_to_absorbance() effectuent ces
conversions.
biodiesel_transm <- absorbance_to_transmittance(biodiesel)
head(biodiesel_transm)
#> wavenumber transmittance sample_id
#> 1 700.7395 84.61941 biodiesel_0
#> 2 702.6032 86.02051 biodiesel_0
#> 3 704.4669 86.42293 biodiesel_0
#> 4 706.3305 87.20593 biodiesel_0
#> 5 708.1942 87.47159 biodiesel_0
#> 6 710.0579 87.77362 biodiesel_0Des fonctions sont fournies pour ajuster la ligne de base des
spectres, ajouter ou soustraire des valeurs scalaires de spectres
entiers, normalisation des spectres, et calculer la moyenne des
spectres, voir : * recalculate_baseline() *
add_scalar_value() et subtract_scalar_value()
* normalize_spectra() * average_spectra()
PlotFTIR peut lire les fichiers de type .csv, .asp, et
.jdx. Le fichier .csv ou .jdx ne doit contenir qu’un seul spectre, avec
des colonnes pour le wavenumber et absorbance
ou transmittance. Les fichiers .asp doivent être conformes
aux spécifications du fichier (non modifiées par l’utilisateur).
irPlotFTIR possède des fonctions pour s’interfacer avec le
paquet ir de Teickner. Ce package offre des capacités de
lignes de base complexes, de lissage, et plus d’outils d’analyse de
données. Plus d’informations sur le paquet ir sont
disponibles dans leur [documetation (via CRAN)] (https://cran.r-project.org/package=ir). Il est également
possible de s’interfacer avec le paquet ChemoSpec de Bryan
Hanson, qui prend en charge les statistiques avancées et la chimiométrie
des données spectrales. Plus d’informations sur [la documentation de
ChemoSpec] (https://bryanhanson.github.io/ChemoSpec/index.html).
Veuillez citer ce paquet dans tout article de journal contenant des images produites à l’aide de ce paquet. Si le paquet est installé à partir de GitHub ou de CRAN, le texte de la date sera correctement rempli avec l’année de publication.
citation("PlotFTIR")
#> To cite package 'PlotFTIR' in publications use:
#>
#> Bulsink P (????). _PlotFTIR: Plot FTIR Spectra_. R package version
#> 1.1.0.9000, <https://github.com/NRCan/PlotFTIR>.
#>
#> A BibTeX entry for LaTeX users is
#>
#> @Manual{,
#> title = {PlotFTIR: Plot FTIR Spectra},
#> author = {Philip Bulsink},
#> note = {R package version 1.1.0.9000},
#> url = {https://github.com/NRCan/PlotFTIR},
#> }Le paquetage a la capacité de changer la langue de l’anglais au
français pour les tracés sur une base individuelle (appeler les
fonctions plot_ftir() avec les arguments
lang = 'en' ou lang = 'fr'). De plus, la
langue par défaut peut être réglée sur l’anglais ou le français en
réglant options('PlotFTIR.lang' = 'en') ou
options('PlotFTIR.lang' = 'fr') respectivement. Ceci peut
être ajouté à votre .RProfile pour persister entre les sessions R.